U-Net con Restricciones Estadísticas de Forma, Aplicada a la Segmentación del Ventrículo Izquierdo en Imágenes de Ecocardiograma
DOI:
https://doi.org/10.17488/RMIB.44.4.10Palabras clave:
análisis estadístico de forma, ecocardiografía, redes neuronales convolucionales, segmentación del ventrículo izquierdoResumen
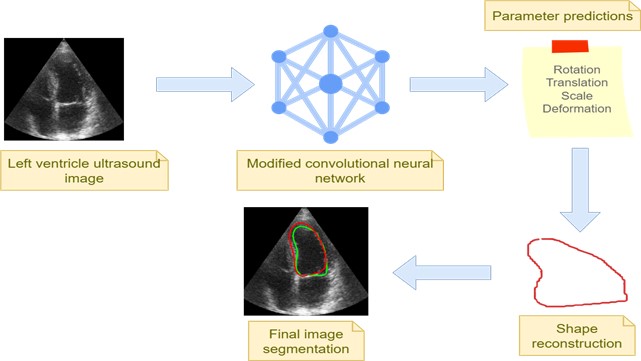
Este artículo tiene como objetivo introducir un enfoque innovador para la segmentación semántica utilizando una red neuronal convolucional (CNN) para predecir los parámetros de forma y posición del ventrículo izquierdo (VI). Nuestro enfoque implica una arquitectura U-Net modificada con una capa de regresión como etapa final, en contraposición a la capa de clasificación tradicional. Esta modificación nos permite predecir todos los parámetros de un modelo estadístico de formas que incluyen rotación, traslación, escala y deformación. La red convolucional se entrena utilizando datos de un modelo de distribución de puntos (PDM) del VI. Los resultados experimentales muestran un coeficiente Dice promedio de 0.82 para imágenes de buena calidad y de 0.66 cuando se incluyen imágenes de calidad media y baja. Nuestro enfoque supera con éxito un problema común en la segmentación semántica basada en CNNs. A diferencia de la clasificación inexacta de píxeles que a menudo conduce a elementos no deseados (blobs), nuestra CNN genera formas estadísticamente válidas. Estas formas tienen un gran potencial para inicializar otros métodos, como los modelos de forma activa (ASMs). En resumen, nuestro enfoque basado en CNN proporciona una solución innovadora para la segmentación semántica, ofreciendo formas y parámetros de posición que pueden mejorar la precisión y confiabilidad de otros métodos de análisis del VI.
Descargas
Citas
N. Paragios and R. Deriche, “Geodesic active regions and level set methods for supervised texture segmentation,” Int. J. Comput. Vis., vol. 46, no. 3, pp. 223–247, Feb. 2002, doi: https://doi.org/10.1023/A:1014080923068
N. Paragios, “A level set approach for shape-driven segmentation and tracking of the left ventricle,” IEEE Trans. Med. Imag., vol. 22, no. 6, pp. 773–776, Jun. 2003, doi: https://doi.org/10.1109/TMI.2003.814785
J. C. Nascimento and J. S. Marques, “Robust shape tracking with multiple models in ultrasound images,” IEEE Trans. Image Process., vol. 17, no. 3, pp. 392–406, Mar. 2008, doi: https://doi.org/10.1109/TIP.2007.915552
V. Zagrodsky, V. Walimbe, C. R. Castro-Pareja, J. X. Qin, J.-M. Song, and R. Shekhar, “Registration-assisted segmentation of real-time 3-D echocardiographic data using deformable models,” IEEE Trans. Med. Imaging., vol. 24, no. 9, pp. 1089–1099, Sep. 2005, doi: https://doi.org/10.1109/tmi.2005.852057
B. Georgescu, X. S. Zhou, D. Comaniciu, and A. Gupta, “Database-guided segmentation of anatomical structures with complex appearance,” in 2005 IEEE Computer Society Conference on Computer Vision and Pattern Recognition (CVPR'05), San Diego, CA, USA, 2005, pp. 429-436 vol. 2, doi: https://doi.org/10.1109/CVPR.2005.119
S. C. Mitchell, B. P. F. Lelieveldt, R. J. van der Geest, H. G. Bosch, J. H. C. Reiber, and M. Sonka, “Multistage hybrid active appearance model matching: Segmentation of left and right ventricles in cardiac MR images,” IEEE Trans. Med. Imag., vol. 20, no. 5, pp. 415–423, May 2001, doi: https://doi.org/10.1109/42.925294
O. Bernard, J. G. Bosch, B. Heyde, M. Alessandrini, et al., “Standardized Evaluation System for Left Ventricular Segmentation Algorithms in 3D Echocardiography,” IEEE Trans. Med. Imaging, vol. 35, no. 4, pp. 967-977, Apr. 2016, doi: https://doi.org/10.1109/TMI.2015.2503890
S. Leclerc, T. Grenier, F. Espinosa and O. Bernard, “A fully automatic and multi-structural segmentation of the left ventricle and the myocardium on highly heterogeneous 2D echocardiographic data,” 2017 IEEE International Ultrasonics Symposium (IUS), Washington, DC, USA, 2017, pp. 1-4, doi: https://doi.org/10.1109/ULTSYM.2017.8092797
T. F. Cootes, C. J. Taylor, D. H. Cooper, J. Graham, “Active shape models their training and application,” Comput. Vis. Image Underst., vol. 61, no. 1, pp. 38–59, Jan. 1995, doi: https://doi.org/10.1006/cviu.1995.1004
Y. Ali, S. Beheshti, F. Janabi-Sharifi, “Echocardiogram segmentation using active shape model and mean squared eigenvalue error,” Biomed. Signal Process. Control, vol. 69, art. no. 102807, Aug. 2021, doi: https://doi.org/10.1016/j.bspc.2021.102807
M. A. Shoaib, J. Huang Chuah, R. Ali, K. Hasikin, et al., “An Overview of Deep Learning Methods for Left Ventricle Segmentation,” Comput. Intell. Neurosci., vol. 2023, art. no. 4208231, 2023, doi: https://doi.org/10.1155/2023/4208231
N. Savioli, M. S. Vieira, P. Lamata, G. Montana, “Automated Segmentation on the Entire Cardiac Cycle Using a Deep Learning Work – Flow,” in 2018 Fifth International Conference on Social Networks Analysis, Management and Security (SNAMS), Valencia, Spain, 2018, pp. 153-158, doi: https://doi.org/10.1109/SNAMS.2018.8554962
X. Zou, Q. Wang, T. Luo, “A novel approach for left ventricle segmentation in tagged MRI,” Comput. Electr. Eng., vol. 95, art. no. 107416, Oct. 2021, doi: https://doi.org/10.1016/j.compeleceng.2021.107416
T. Wech, M. J. Ankenbrand, T. A. Bley, J. F. Heidenreich, “A data-driven semantic segmentation model for direct cardiac functional analysis based on undersampled radial MR cine series,” Magn. Reason. Med., vol. 87, no. 2, pp. 972-983, Feb. 2022, doi: https://doi.org/10.1002/mrm.29017
G. Veni, M. Moradi, H. Bulu, G. Narayan, and T. Syeda-Mahmood, “Echocardiography segmentation based on a shape-guided deformable model driven by a fully convolutional network prior,” 2018 IEEE 15th International Symposium on Biomedical Imaging (ISBI 2018), Washington, DC, USA, 2018, pp. 898-902, doi: https://doi.org/10.1109/ISBI.2018.8363716
W.-Y. Hsu, “Automatic Left Ventricle Recognition, Segmentation and Tracking in Cardiac Ultrasound Image Sequences,” IEEE Access, vol. 7, pp. 140524-140533, 2019, doi: https://doi.org/10.1109/ACCESS.2019.2920957
Y. Li, W. Lu, P. Monkam, Z. Zhu, W. Wu, M. Liu, “LVSnake: Accurate and robust left ventricle contour localization for myocardial infarction detection,” Biomed. Signal Process. Control, vol. 85, art. no. 105076, Aug. 2023, doi: https://doi.org/10.1016/j.bspc.2023.105076
S. Peng, W. Jiang, H. Pi, X. Li, H. Bao, and X. Zhou, “Deep Snake for Real-Time Instance Segmentation,” 2020, arXiv: 2001.01629, doi:
https://doi.org/10.48550/arXiv.2001.01629
O. Ronneberger, P. Fischer, T. Brox, “U-Net: Convolutional Networks for Biomedical Image Segmentation,” Medical Image Computing and Computer-Assisted Intervention – MICCAI 2015, Munich, Germany, 2015, pp. 234-241, doi: https://doi.org/10.1007/978-3-319-24574-4_28
S. Leclerc, E. Smistad, J. Pedrosa, A. Østvik, et al., “Deep Learning for Segmentation using an Open Large-Scale Dataset in 2D Echocardiography,” IEEE Trans. Med. Imaging, vol. 38, no. 9, pp. 2198-2210, Sep. 2019, doi: https://doi.org/10.1109/TMI.2019.2900516
EchoNet-Dynamic: a Large New Cardiac Motion Video Data Resource for Medical Machine Learning, github, 2019. [Online]. Available: https://echonet.github.io/dynamic/index.html#motivation
E. Galicia Gomez, F. Torres Robles, B. Escalante Rmirez, J. Olveres, F. Arámbula Cosío, “Full multi resolution active shape model for left ventricle segmentation,” in 17th International Symposium on Medical Information Processing and Analysis, Campinas, Brazil, 2021, doi: https://doi.org/10.1117/12.2606252
M. B. Stegmann, D. Delgado Gomez. (2002). A Brief Introduction to Statistical Shape Analysis [Online]. Available: http://www2.imm.dtu.dk/pubdb/edoc/imm403.pdf
A. Cervantes-Guzmán, K. McPherson, J. Olveres, C. F. Moreno-García, F. Torres Robles, E. Elyan, B. Escalante-Ramírez, “Robust cardiac segmentation corrected with heuristics,” PLoS One, vol. 18, no. 10, art. no. E0293560, Oct. 2023, doi: https://doi.org/10.1371/journal.pone.0293560
Descargas
Publicado
Cómo citar
Número
Sección
Licencia
Derechos de autor 2023 Revista Mexicana de Ingenieria Biomedica

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial 4.0.
Una vez que el artículo es aceptado para su publicación en la RMIB, se les solicitará al autor principal o de correspondencia que revisen y firman las cartas de cesión de derechos correspondientes para llevar a cabo la autorización para la publicación del artículo. En dicho documento se autoriza a la RMIB a publicar, en cualquier medio sin limitaciones y sin ningún costo. Los autores pueden reutilizar partes del artículo en otros documentos y reproducir parte o la totalidad para su uso personal siempre que se haga referencia bibliográfica al RMIB. No obstante, todo tipo de publicación fuera de las publicaciones académicas del autor correspondiente o para otro tipo de trabajos derivados y publicados necesitaran de un permiso escrito de la RMIB.











