Evaluación del Rendimiento de Métodos de Transformación de Series Temporales Biomédicas para Tareas de Clasificación
DOI:
https://doi.org/10.17488/RMIB.44.4.7Palabras clave:
clasificación, datos biométricos, redes neuronales convolucionales, series temporales, transformacionesResumen
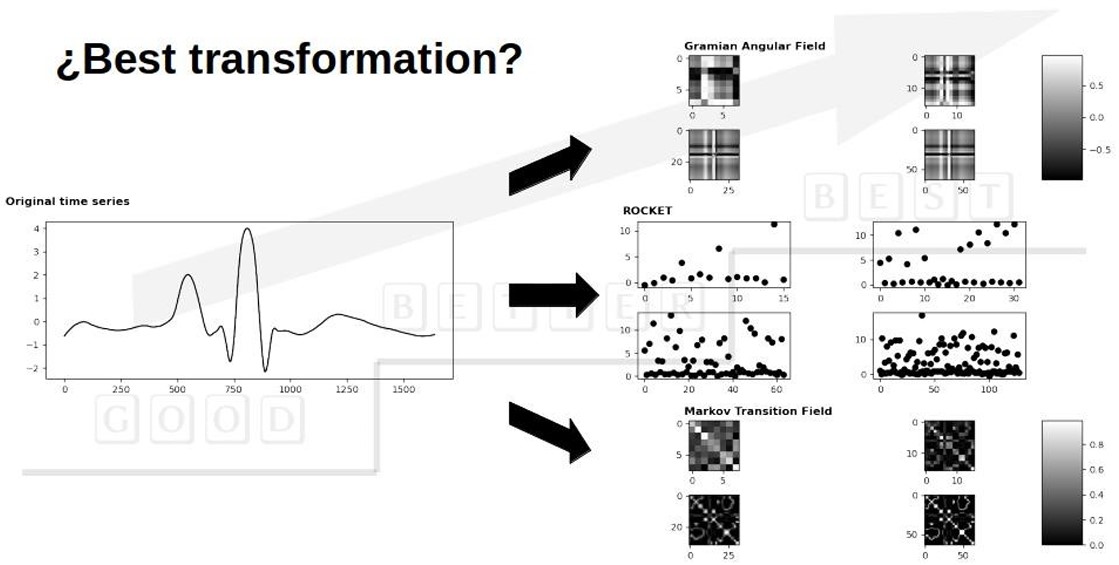
La extracción de características de series temporales es esencial en diversos campos, pero sigue siendo un desafío. Por lo tanto, es crucial identificar métodos apropiados capaces de extraer información pertinente que pueda mejorar significativamente el rendimiento de clasificación. Entre estos métodos se encuentran aquellos que traducen las series temporales a diferentes dominios. Este estudio investiga tres enfoques distintos de transformación de series temporales para abordar los desafíos de clasificación de series temporales en datos biomédicos. El primer método implica una transformación de vector de respuesta, mientras que los otros dos emplean técnicas de transformación de imagen: RandOm Convolutional KErnel Transform (ROCKET), Gramian Angular Fields y Markov Transition Fields. Estos métodos de transformación se aplicaron a cinco conjuntos de datos biomédicos, explorando diversas configuraciones de formato para determinar la técnica y configuración de representación óptima para la entrada, lo que a su vez mejora el rendimiento de clasificación. Se realizaron evaluaciones sobre la efectividad de estos métodos en conjunción con dos algoritmos de clasificación. Los resultados subrayan la importancia de estas técnicas de transformación de series temporales como facilitadoras para mejorar los algoritmos de clasificación documentados en la literatura actual.
Descargas
Citas
M. Middlehurst, P. Schäfer, and A. Bagnall, “Bake off redux: a review and experimental evaluation of recent time series classification algorithms,” 2023, arXiv:2304.13029, doi: https://doi.org/10.48550/arXiv.2304.13029
H. Ismail Fawaz, G. Forestier, J. Weber, L. Idoumghar, and P.-A. Muller, “Deep learning for time series classification: a review,” Data Min. Knowl. Discov., vol. 33, no. 4, pp. 917–963, 2019, doi: https://doi.org/10.1007/s10618-019-00619-1
C. Li, J. Xiong, X. Zhu, Q. Zhang, and S. Wang, “Fault Diagnosis Method Based on Encoding Time Series and Convolutional Neural Network,” IEEE Access, vol. 8, pp. 165232–165246, 2020, doi: https://doi.org/10.1109/ACCESS.2020.3021007
G. R. Garcia, G. Michau, M. Ducoffe, J. Sen Gupta, and O. Fink, “Temporal signals to images: Monitoring the condition of industrial assets with deep learning image processing algorithms,” Proc. Inst. Mech. Eng. O J. Risk Reliab., vol. 236, no. 4, pp. 617–627, 2022, doi: https://doi.org/10.1177/1748006X21994446
J. Lines, S. Taylor, and A. Bagnall, “Hive-cote: The hierarchical vote collective of transformation-based ensembles for time series classification,” in 2016 IEEE 16th international conference on data mining (ICDM), Barcelona, Spain, 2016, pp. 1041–1046, doi: https://doi.org/10.1109/ICDM.2016.0133
Z. Wang, W. Yan, and T. Oates, “Time series classification from scratch with deep neural networks: A strong baseline,” in 2017 International Joint Conference on Neural Networks (IJCNN), Anchorage, AK, USA, 2017, pp. 1578–1585, doi: https://doi.org/10.1109/IJCNN.2017.7966039
H. Ismail Fawaz, B. Lucas, G. Forestier, C. Pelletier, et al., “Inceptiontime: Finding alexnet for time series classification,” Data Min. Knowl. Discov., vol. 34, no. 6, pp. 1936–1962, 2020, doi: https://doi.org/10.1007/s10618-020-00710-y
H. A. Dau, A. Bagnall, K. Kamgar, C.-C. M. Yeh, Y. Zhu, S. Gharghabi, C. A. Ratanamahatana, E. Keogh, “The UCR time series archive,” IEEE/CAA J. Autom. Sin., vol. 6, no. 6, pp. 1293–1305, 2019, doi: https://doi.org/10.1109/JAS.2019.1911747
A. Dempster, F. Petitjean, and G. I. Webb, “ROCKET: exceptionally fast and accurate time series classification using random convolutional kernels,” Data Min. Knowl. Discov., vol. 34, no. 5, pp. 1454–1495, 2020, doi: https://doi.org/10.1007/s10618-020-00701-z
Z. Wang and T. Oates, “Imaging time-series to improve classification and imputation,” 2015, arXiv:1506.00327, 2015, doi: https://doi.org/10.48550/arXiv.1506.00327
J. Faouzi and H. Janati, “pyts: A Python Package for Time Series Classification,” J. Mach. Learn. Res., vol. 21, no. 46, pp. 1–6, 2020. [Online]. Available: http://jmlr.org/papers/v21/19-763.html
F. Chollet, “Keras.” 2015, GitHub. [Online]. Available: https://github.com/fchollet/keras
F. Pedregosa, G. Varoquaux, A. Gramfort, V. Michel, et al., “Scikit-learn: Machine learning in Python,” J. Mach. Learn. Res., vol. 12, no. 85, 2825-2830. [Online]. Available: http://www.jmlr.org/papers/v12/pedregosa11a.html
Tanu and D. Kakkar, “Accounting for order-frame length tradeoff of Savitzky-Golay smoothing filters,” in 2018 5th International Conference on Signal Processing and Integrated Networks (SPIN), Noida, India, 2018, pp. 805–810, doi: https://doi.org/10.1109/SPIN.2018.8474194
X. Li, Y. Kang, and F. Li, “Forecasting with time series imaging,” Expert. Syst. Appl., vol. 160, art. no. 113680, 2020, doi: https://doi.org/10.1016/j.eswa.2020.113680
A. M. Gonzalez-Zapata, L. G. de la Fraga, B. Ovilla-Martinez, E. Tlelo-Cuautle, and I. Cruz-Vega, “Enhanced FPGA implementation of Echo State Networks for chaotic time series prediction,” Integration, vol. 92, pp. 48–57, 2023, doi: https://doi.org/10.1016/j.vlsi.2023.05.002
A. Ben Said and A. Erradi, “Deep-Gap: A deep learning framework for forecasting crowdsourcing supply-demand gap based on imaging time series and residual learning,” 2019, arXiv:1911.07625, doi: https://doi.org/10.48550/arXiv.1911.07625
W. Ni, C. Zhang, T. Liu, Q. Zeng, L. Xu, and H. Wang, “An efficient astronomical seeing forecasting method by random convolutional Kernel transformation,” Eng. Appl. Artif. Intell., vol. 127, art. no. 107259, 2024, doi: https://doi.org/10.1016/j.engappai.2023.107259
J. Marco-Blanco and R. Cuevas, “Time Series Clustering With Random Convolutional Kernels,” 2023, arXiv:2305.10457, doi: https://doi.org/10.48550/arXiv.2305.10457
B. Dhariyal, T. Le Nguyen, and G. Ifrim, “Back to Basics: A Sanity Check on Modern Time Series Classification Algorithms,” 2023, arXiv:2308.07886, doi: https://doi.org/10.48550/arXiv.2308.07886
M. Middlehurst, J. Large, M. Flynn, J. Lines, A. Bostrom, and A. Bagnall, “HIVE-COTE 2.0: a new meta ensemble for time series classification,” Mach. Learn., vol. 110, no. 11–12, pp. 3211–3243, 2021, doi: https://doi.org/10.1007/s10994-021-06057-9
Descargas
Publicado
Cómo citar
Número
Sección
Licencia
Derechos de autor 2023 Revista Mexicana de Ingenieria Biomedica

Esta obra está bajo una licencia internacional Creative Commons Atribución-NoComercial 4.0.
Una vez que el artículo es aceptado para su publicación en la RMIB, se les solicitará al autor principal o de correspondencia que revisen y firman las cartas de cesión de derechos correspondientes para llevar a cabo la autorización para la publicación del artículo. En dicho documento se autoriza a la RMIB a publicar, en cualquier medio sin limitaciones y sin ningún costo. Los autores pueden reutilizar partes del artículo en otros documentos y reproducir parte o la totalidad para su uso personal siempre que se haga referencia bibliográfica al RMIB. No obstante, todo tipo de publicación fuera de las publicaciones académicas del autor correspondiente o para otro tipo de trabajos derivados y publicados necesitaran de un permiso escrito de la RMIB.











